| Full name: microtubule associated protein 2 | Alias Symbol: MAP2A|MAP2B|MAP2C | ||
| Type: protein-coding gene | Cytoband: 2q34 | ||
| Entrez ID: 4133 | HGNC ID: HGNC:6839 | Ensembl Gene: ENSG00000078018 | OMIM ID: 157130 |
| Drug and gene relationship at DGIdb | |||
Expression of MAP2:
| Dataset | Gene | EntrezID | Probe | Log2FC | Adj.pValue | Expression |
|---|---|---|---|---|---|---|
| GSE17351 | MAP2 | 4133 | 225540_at | -1.2818 | 0.1365 | |
| GSE20347 | MAP2 | 4133 | 210015_s_at | 0.3020 | 0.3382 | |
| GSE23400 | MAP2 | 4133 | 210015_s_at | 0.1092 | 0.3406 | |
| GSE26886 | MAP2 | 4133 | 225540_at | -0.5799 | 0.3754 | |
| GSE29001 | MAP2 | 4133 | 210015_s_at | -0.5770 | 0.2010 | |
| GSE38129 | MAP2 | 4133 | 210015_s_at | 0.3503 | 0.2245 | |
| GSE45670 | MAP2 | 4133 | 225540_at | -0.3642 | 0.5041 | |
| GSE53622 | MAP2 | 4133 | 32161 | -0.5381 | 0.0063 | |
| GSE53624 | MAP2 | 4133 | 32161 | -0.5355 | 0.0002 | |
| GSE63941 | MAP2 | 4133 | 225540_at | -1.5876 | 0.3640 | |
| GSE77861 | MAP2 | 4133 | 225540_at | 0.8796 | 0.3389 | |
| GSE97050 | MAP2 | 4133 | A_33_P3277913 | 0.0912 | 0.6496 | |
| SRP007169 | MAP2 | 4133 | RNAseq | 0.9992 | 0.1091 | |
| SRP008496 | MAP2 | 4133 | RNAseq | 0.9278 | 0.0897 | |
| SRP064894 | MAP2 | 4133 | RNAseq | -0.8571 | 0.0191 | |
| SRP133303 | MAP2 | 4133 | RNAseq | 0.0550 | 0.9224 | |
| SRP159526 | MAP2 | 4133 | RNAseq | 0.7674 | 0.1919 | |
| SRP193095 | MAP2 | 4133 | RNAseq | 0.2329 | 0.5397 | |
| SRP219564 | MAP2 | 4133 | RNAseq | -0.4886 | 0.3772 | |
| TCGA | MAP2 | 4133 | RNAseq | -0.0126 | 0.9527 |
Upregulated datasets: 0; Downregulated datasets: 0.
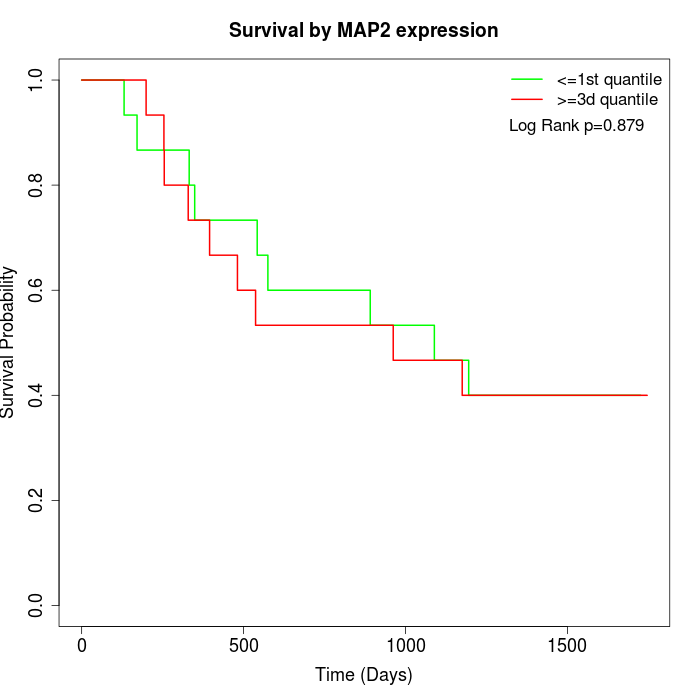
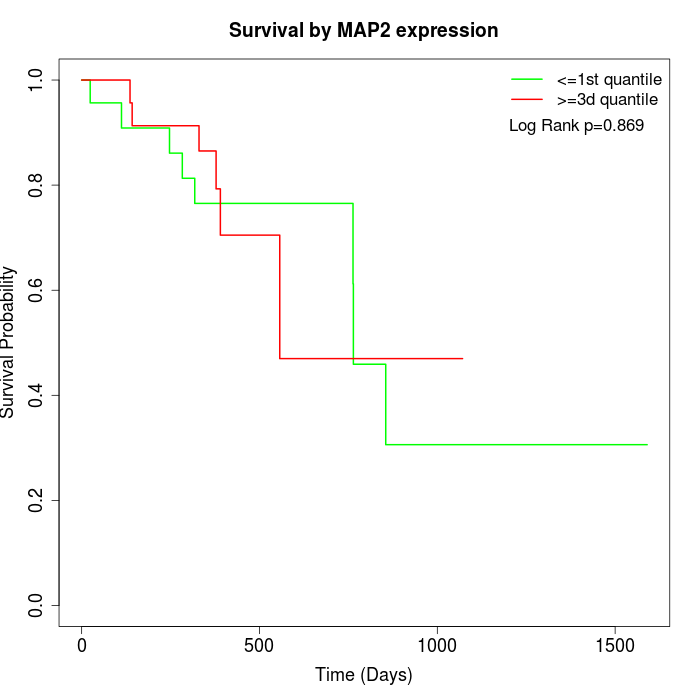
Survival by MAP2 expression:
Note: Click image to view full size file.
Copy number change of MAP2:
| Dataset | Gene | EntrezID | Gain | Loss | Normal | Detail |
|---|---|---|---|---|---|---|
| GSE15526 | MAP2 | 4133 | 3 | 11 | 16 | |
| GSE20123 | MAP2 | 4133 | 3 | 11 | 16 | |
| GSE43470 | MAP2 | 4133 | 1 | 6 | 36 | |
| GSE46452 | MAP2 | 4133 | 0 | 5 | 54 | |
| GSE47630 | MAP2 | 4133 | 4 | 5 | 31 | |
| GSE54993 | MAP2 | 4133 | 0 | 3 | 67 | |
| GSE54994 | MAP2 | 4133 | 9 | 9 | 35 | |
| GSE60625 | MAP2 | 4133 | 0 | 3 | 8 | |
| GSE74703 | MAP2 | 4133 | 1 | 5 | 30 | |
| GSE74704 | MAP2 | 4133 | 2 | 6 | 12 | |
| TCGA | MAP2 | 4133 | 12 | 24 | 60 |
Total number of gains: 35; Total number of losses: 88; Total Number of normals: 365.
Somatic mutations of MAP2:
Generating mutation plots.
Highly correlated genes for MAP2:
Showing top 20/24 corelated genes with mean PCC>0.5.
| Gene1 | Gene2 | Mean PCC | Num. Datasets | Num. PCC<0 | Num. PCC>0.5 |
|---|---|---|---|---|---|
| MAP2 | MAMLD1 | 0.772694 | 3 | 0 | 3 |
| MAP2 | GSTM5 | 0.724616 | 3 | 0 | 3 |
| MAP2 | DCBLD2 | 0.699836 | 3 | 0 | 3 |
| MAP2 | SCAMP5 | 0.685947 | 3 | 0 | 3 |
| MAP2 | SYPL2 | 0.682582 | 3 | 0 | 3 |
| MAP2 | CUTC | 0.640774 | 4 | 0 | 3 |
| MAP2 | FAT4 | 0.636166 | 3 | 0 | 3 |
| MAP2 | TCEAL7 | 0.617549 | 3 | 0 | 3 |
| MAP2 | ARNTL | 0.600983 | 3 | 0 | 3 |
| MAP2 | GCLM | 0.587662 | 8 | 0 | 6 |
| MAP2 | OSGIN1 | 0.585761 | 9 | 0 | 6 |
| MAP2 | CYP4F11 | 0.559291 | 9 | 0 | 7 |
| MAP2 | SCN9A | 0.558946 | 8 | 0 | 4 |
| MAP2 | INPP4B | 0.54985 | 5 | 0 | 3 |
| MAP2 | CYP4F3 | 0.545368 | 11 | 0 | 7 |
| MAP2 | G6PD | 0.543472 | 8 | 0 | 5 |
| MAP2 | CCNT2 | 0.539458 | 4 | 0 | 3 |
| MAP2 | ANXA10 | 0.532678 | 8 | 0 | 4 |
| MAP2 | NR0B1 | 0.526939 | 8 | 0 | 5 |
| MAP2 | HSPB3 | 0.523734 | 4 | 0 | 3 |
For details and further investigation, click here