| Full name: src kinase associated phosphoprotein 2 | Alias Symbol: RA70|SKAP-HOM|SKAP55R|SAPS | ||
| Type: protein-coding gene | Cytoband: 7p15.2 | ||
| Entrez ID: 8935 | HGNC ID: HGNC:15687 | Ensembl Gene: ENSG00000005020 | OMIM ID: 605215 |
| Drug and gene relationship at DGIdb | |||
Screen Evidence:
| |||
Expression of SKAP2:
| Dataset | Gene | EntrezID | Probe | Log2FC | Adj.pValue | Expression |
|---|---|---|---|---|---|---|
| GSE17351 | SKAP2 | 8935 | 204362_at | -0.6158 | 0.1726 | |
| GSE20347 | SKAP2 | 8935 | 204362_at | -0.3252 | 0.0763 | |
| GSE23400 | SKAP2 | 8935 | 204362_at | -0.0575 | 0.5052 | |
| GSE26886 | SKAP2 | 8935 | 204362_at | -0.5042 | 0.0592 | |
| GSE29001 | SKAP2 | 8935 | 204362_at | -0.1852 | 0.7064 | |
| GSE38129 | SKAP2 | 8935 | 204362_at | -0.3242 | 0.0547 | |
| GSE45670 | SKAP2 | 8935 | 204362_at | -0.1908 | 0.1619 | |
| GSE53622 | SKAP2 | 8935 | 49973 | -0.8297 | 0.0000 | |
| GSE53624 | SKAP2 | 8935 | 49973 | -0.6866 | 0.0000 | |
| GSE63941 | SKAP2 | 8935 | 204362_at | -0.9916 | 0.1478 | |
| GSE77861 | SKAP2 | 8935 | 225639_at | -0.8722 | 0.0274 | |
| GSE97050 | SKAP2 | 8935 | A_33_P3388312 | -1.0370 | 0.0830 | |
| SRP007169 | SKAP2 | 8935 | RNAseq | -1.0166 | 0.0020 | |
| SRP008496 | SKAP2 | 8935 | RNAseq | -0.5296 | 0.0077 | |
| SRP064894 | SKAP2 | 8935 | RNAseq | -0.2614 | 0.1966 | |
| SRP133303 | SKAP2 | 8935 | RNAseq | 0.0582 | 0.7627 | |
| SRP159526 | SKAP2 | 8935 | RNAseq | -0.7059 | 0.0668 | |
| SRP193095 | SKAP2 | 8935 | RNAseq | -0.6233 | 0.0000 | |
| SRP219564 | SKAP2 | 8935 | RNAseq | -0.5125 | 0.2813 | |
| TCGA | SKAP2 | 8935 | RNAseq | 0.1052 | 0.1195 |
Upregulated datasets: 0; Downregulated datasets: 1.
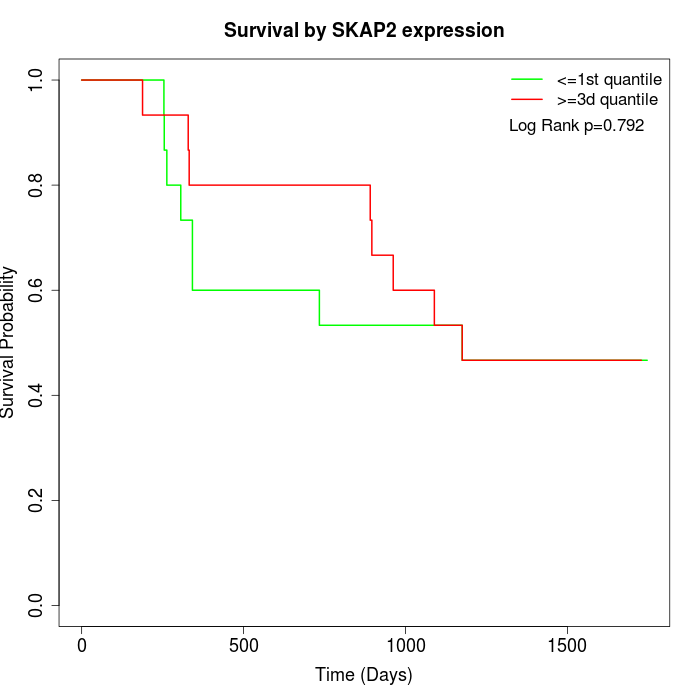
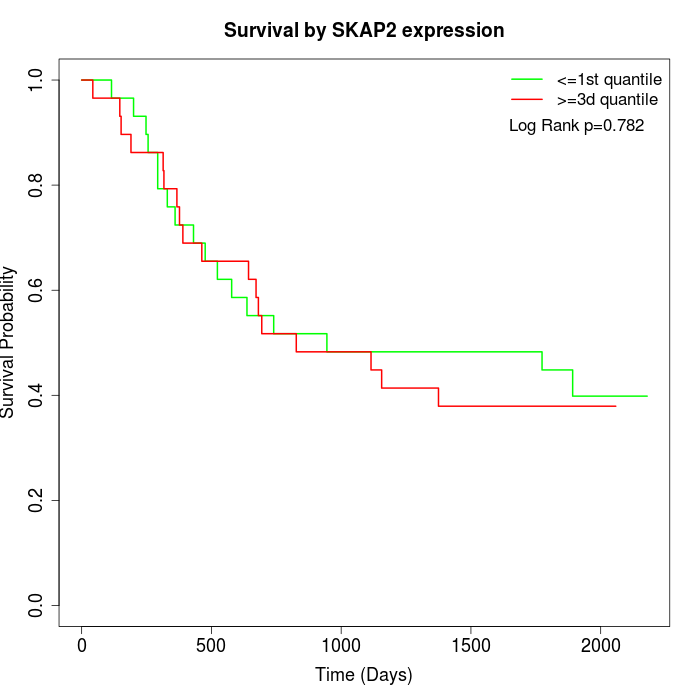
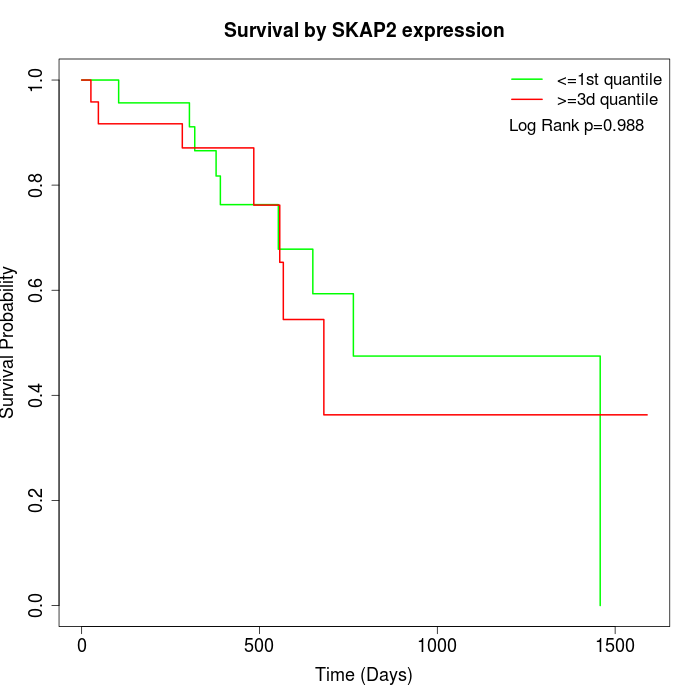
Survival by SKAP2 expression:
Note: Click image to view full size file.
Copy number change of SKAP2:
| Dataset | Gene | EntrezID | Gain | Loss | Normal | Detail |
|---|---|---|---|---|---|---|
| GSE15526 | SKAP2 | 8935 | 16 | 1 | 13 | |
| GSE20123 | SKAP2 | 8935 | 16 | 1 | 13 | |
| GSE43470 | SKAP2 | 8935 | 4 | 0 | 39 | |
| GSE46452 | SKAP2 | 8935 | 10 | 2 | 47 | |
| GSE47630 | SKAP2 | 8935 | 9 | 1 | 30 | |
| GSE54993 | SKAP2 | 8935 | 0 | 6 | 64 | |
| GSE54994 | SKAP2 | 8935 | 21 | 2 | 30 | |
| GSE60625 | SKAP2 | 8935 | 0 | 0 | 11 | |
| GSE74703 | SKAP2 | 8935 | 4 | 0 | 32 | |
| GSE74704 | SKAP2 | 8935 | 10 | 1 | 9 | |
| TCGA | SKAP2 | 8935 | 54 | 7 | 35 |
Total number of gains: 144; Total number of losses: 21; Total Number of normals: 323.
Somatic mutations of SKAP2:
Generating mutation plots.
Highly correlated genes for SKAP2:
Showing top 20/993 corelated genes with mean PCC>0.5.
| Gene1 | Gene2 | Mean PCC | Num. Datasets | Num. PCC<0 | Num. PCC>0.5 |
|---|---|---|---|---|---|
| SKAP2 | PPWD1 | 0.799483 | 4 | 0 | 4 |
| SKAP2 | PHYHIPL | 0.799132 | 3 | 0 | 3 |
| SKAP2 | KIN | 0.762431 | 3 | 0 | 3 |
| SKAP2 | UBQLN1 | 0.741251 | 3 | 0 | 3 |
| SKAP2 | TMEM30A | 0.737087 | 4 | 0 | 3 |
| SKAP2 | ZNF33A | 0.735975 | 3 | 0 | 3 |
| SKAP2 | CLEC3B | 0.732014 | 3 | 0 | 3 |
| SKAP2 | THUMPD3 | 0.728399 | 3 | 0 | 3 |
| SKAP2 | SNN | 0.728302 | 3 | 0 | 3 |
| SKAP2 | HPS5 | 0.722198 | 3 | 0 | 3 |
| SKAP2 | MTIF3 | 0.718546 | 5 | 0 | 5 |
| SKAP2 | BDP1 | 0.712314 | 4 | 0 | 4 |
| SKAP2 | RSRC2 | 0.712239 | 4 | 0 | 3 |
| SKAP2 | HSPA2 | 0.711689 | 4 | 0 | 3 |
| SKAP2 | INTS12 | 0.710334 | 3 | 0 | 3 |
| SKAP2 | RNF185 | 0.70946 | 4 | 0 | 4 |
| SKAP2 | GPBP1 | 0.704814 | 4 | 0 | 4 |
| SKAP2 | AKIRIN1 | 0.703026 | 3 | 0 | 3 |
| SKAP2 | CRNKL1 | 0.699748 | 3 | 0 | 3 |
| SKAP2 | SORCS1 | 0.699242 | 5 | 0 | 5 |
For details and further investigation, click here